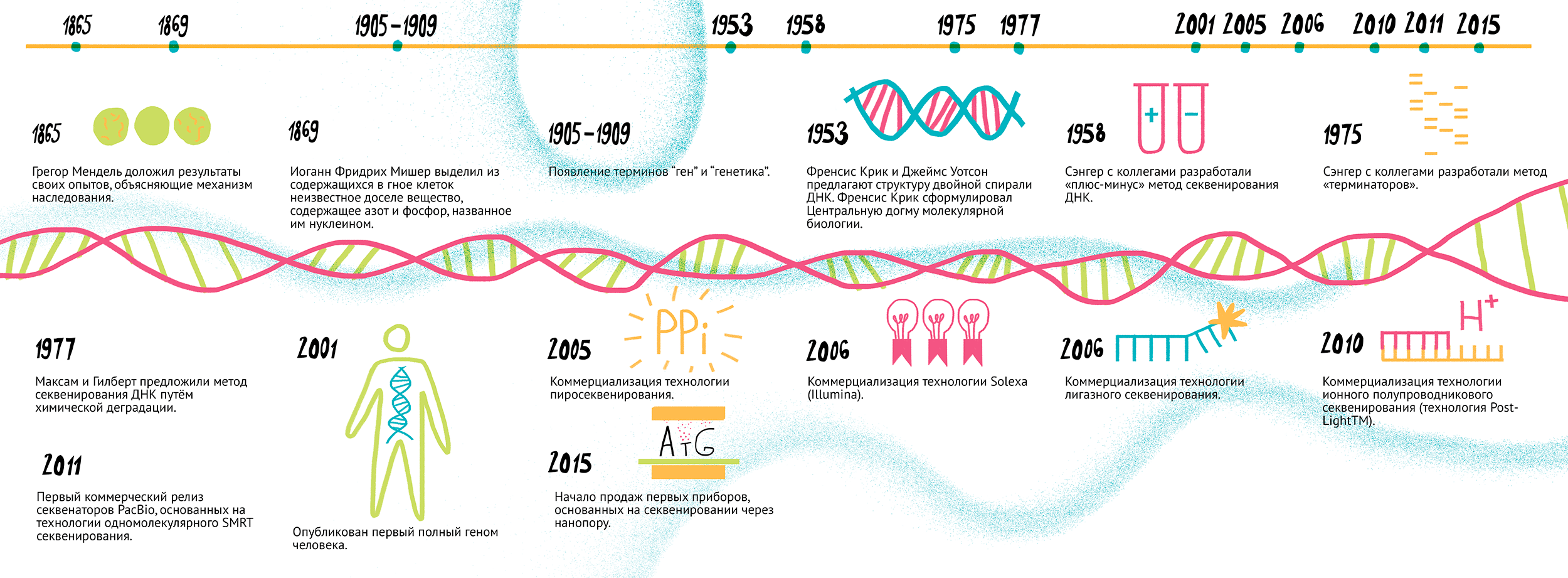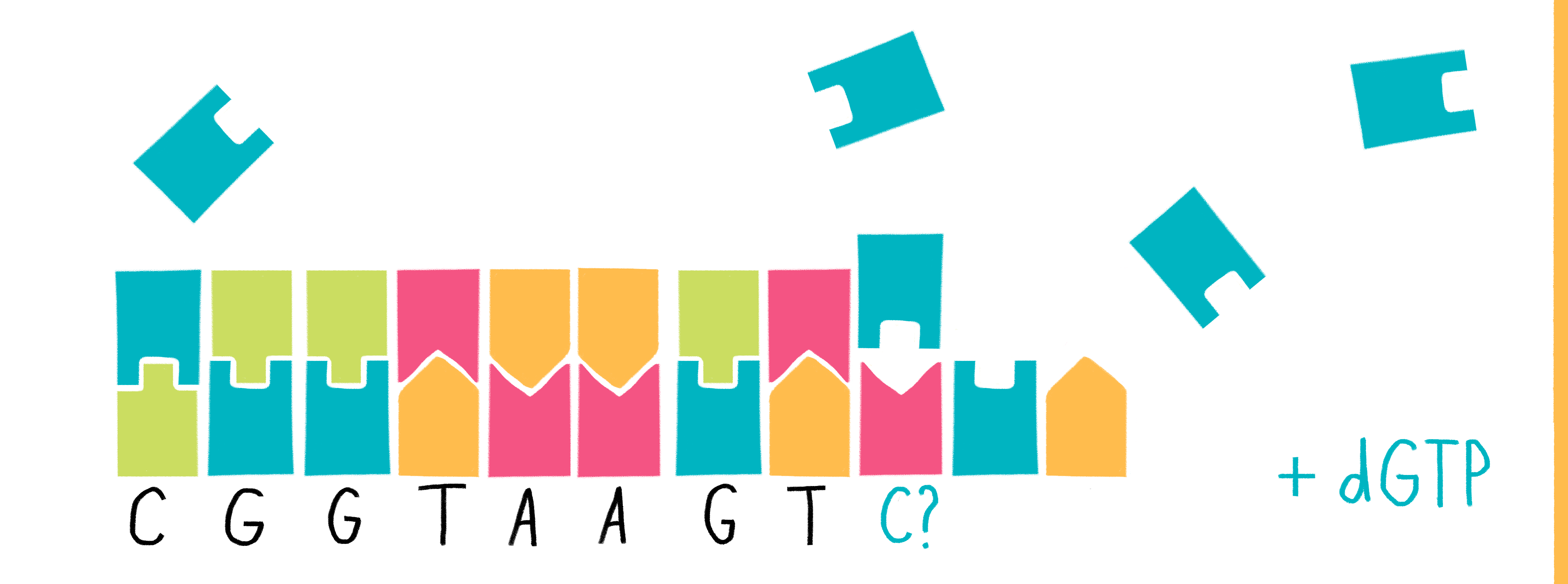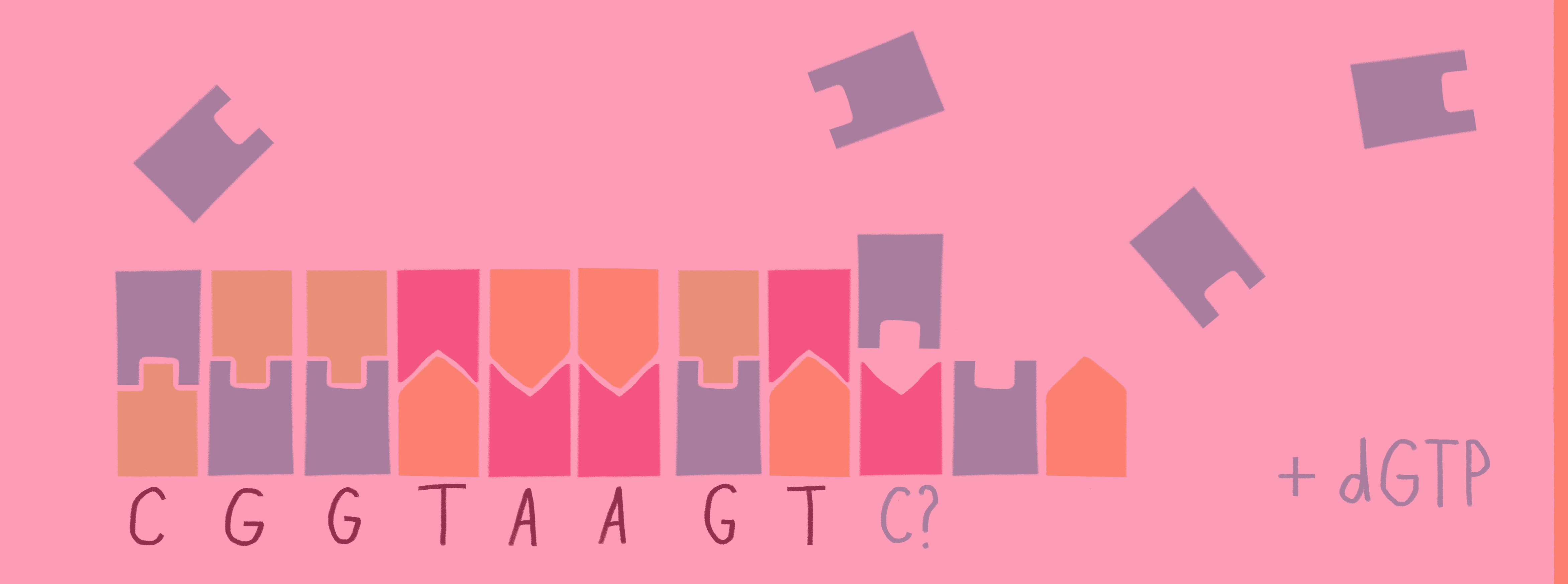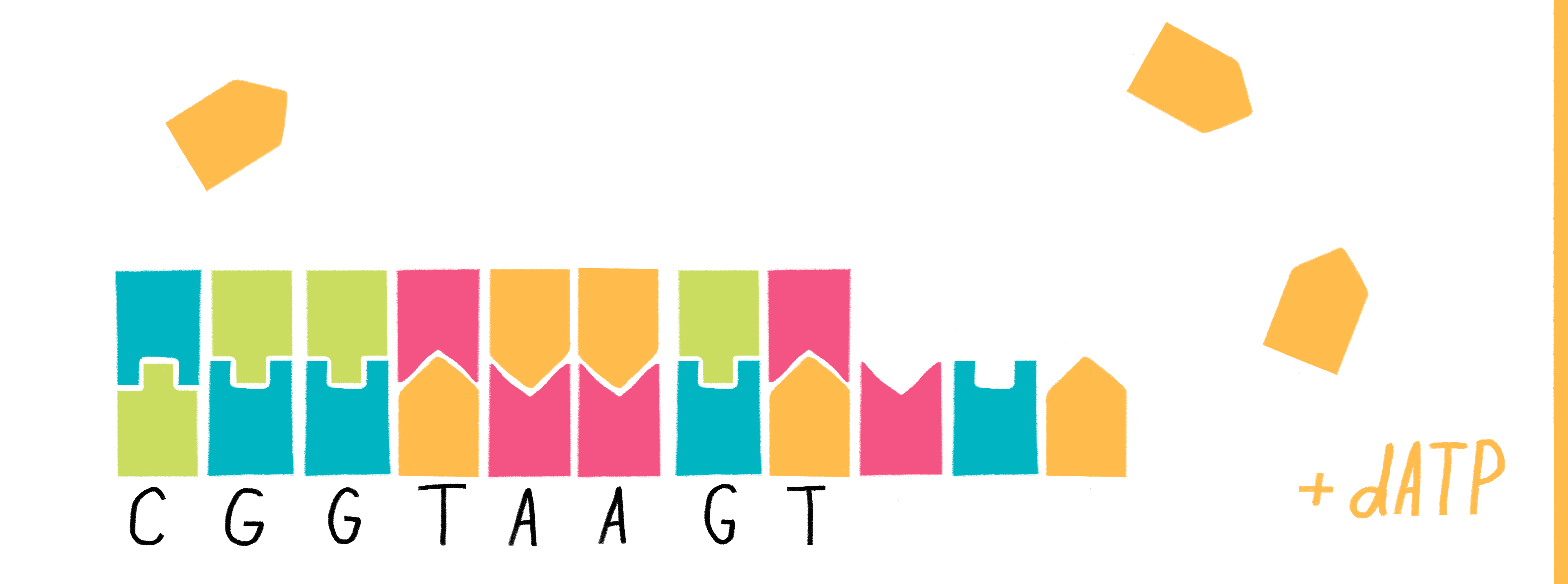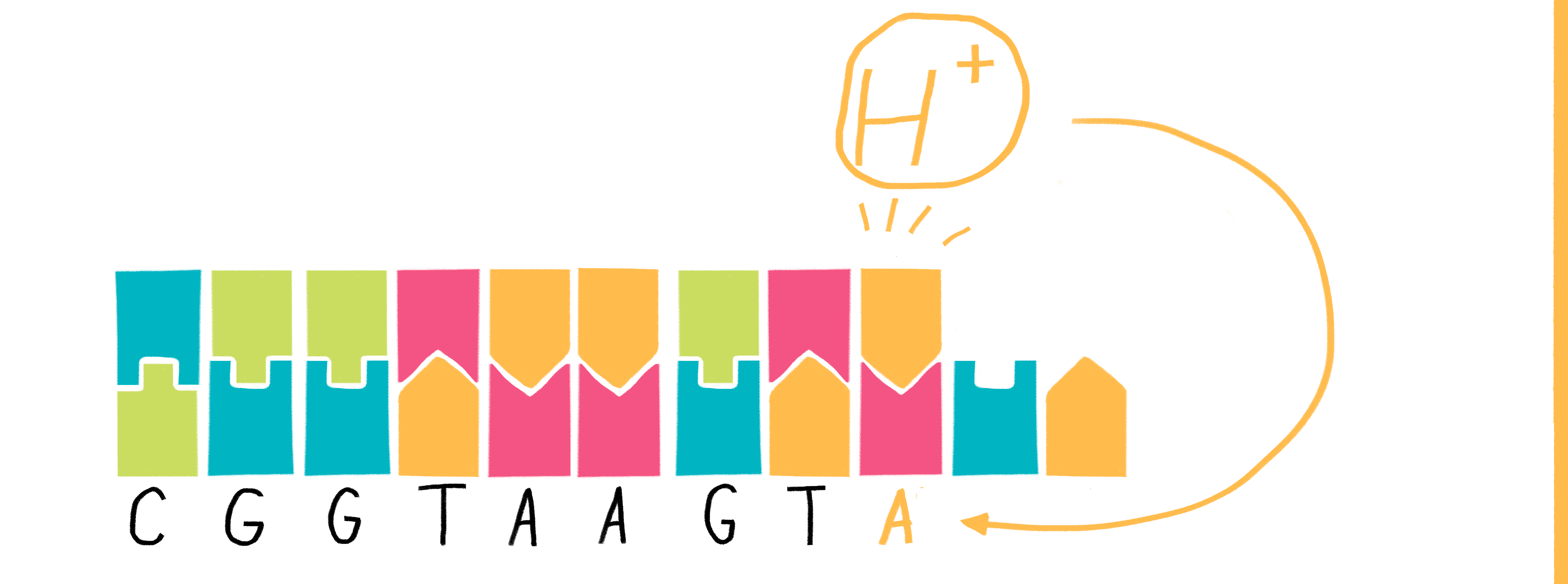Секвенирование что это значит
Секвенирование геномов для «чайников»
 Геномика: постановка задачи и методы секвенирования
Геномика: постановка задачи и методы секвенирования
Сергей Николенко, кандидат физико-математических наук, старший научный сотрудник лаборатории вычислительной биологии Санкт-Петербургского Академического Университета в серии статей говорит о некоторых задачах биоинформатики, связанных со сборкой и анализом геномов, делая акцент на математической, комбинаторной постановке задачи. В данном, вводном, тексте речь идет о том, как выглядят входные данные для сборки геномов и как их получают.
Как выглядит молекула ДНК?
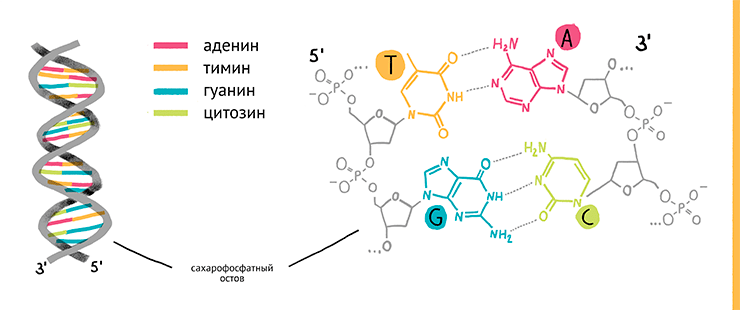
Начнем с того, как выглядит молекула ДНК. Молекулы полимеров характеризуются первичной структурой, под которой понимается просто состав молекулы (в данном случае – последовательность букв A, C, G и T, которые и составляют геном), вторичной структурой, т.е. тем, какие именно химические связи устанавливаются между этими компонентами и какие в результате получаются базовые пространственные структуры (в данном случае – двойная спираль), и третичной структурой, т.е. тем, как вторичная структура «уложена» в пространстве. Вторичная структура ДНК представляет собой двойную спираль, состоящую из четырёх разных нуклеотидов.

Рисунок из Википедии
Нуклеотиды обозначаются по содержащимся в них азотистым основаниям: аденину (A), цитозину (C), гуанину (G) и тимину (T) (есть ещё урацил, который в РНК заменяет тимин), и в дальнейшем мы всегда будем пользоваться этими буквами. В двойной спирали эти нуклеотиды связаны друг с другом водородными связями, и связь устанавливается по принципу комплементарности: если в одной нити ДНК стоит A, то в комплементарной нити будет T, а если в одной нити C, то в другой будет G. Именно это позволяет относительно просто проводить репликацию (копирование) ДНК, например, при делении клетки: для этого достаточно просто разорвать водородные связи, разделив двойную спираль на нити, после чего парная нить для каждого «потомка» автоматически соберётся правильно. Важно понять, что ДНК – это две копии одного и того же «текста» из четырёх «букв»; «буквы» в копиях не идентичны, но однозначно соответствуют друг другу. Например:
Было бы, конечно, удобно, если бы нам удалось аккуратно «вытянуть» одну нить ДНК и спокойно, нуклеотид за нуклеотидом, «прочесть» эту нить от начала до конца. При таком, идеальном, методе секвенирования (чтения ДНК) никаких хитрых алгоритмов не понадобилось бы. К сожалению, на данном этапе такое невозможно, и приходится довольствоваться результатами того секвенирования, которое есть.
Что такое секвенирование?
Секвенирование (sequencing) – это общее название методов, которые позволяют установить последовательность нуклеотидов в молекуле ДНК. В настоящее время нет ни одного метода секвенирования, который бы работал для молекулы ДНК целиком; все они устроены так: сначала готовится большое число небольших участков ДНК (клонируется молекула ДНК многократно и «разрезается» в случайных местах), а потом читается каждый участок по отдельности.
Клонирование происходит либо просто выращиванием клеток в чашке Петри, либо (в случаях, когда это было бы слишком медленно или по каким-то причинам не получилось бы) при помощи так называемой полимеразной цепной реакции. В кратком и неточном изложении работает она примерно так: сначала ДНК денатурируют, т.е. разрушают водородные связи, получая отдельные нити. Затем к ДНК присоединяют так называемые праймеры; это короткие участки ДНК, к которым может присоединиться ДНК-полимераза – соединение, которое, собственно, и занимается копированием (репликацией) нити ДНК.

Рисунок из Википедии
На следующем этапе полимераза копирует ДНК, после чего процесс можно повторять: после новой денатурации отдельных нитей будет уже вдвое больше, на третьем цикле – вчетверо, и так далее.
Все эти эффекты достигаются в основном с помощью изменений температуры смеси из ДНК, праймеров и полимеразы; для наших целей важно, что это достаточно точный процесс, и ошибки в нём редки, а на выходе получается большое число копий участков одной и той же ДНК. Разные методы секвенирования отличаются друг от друга не методами клонирования, а тем, как потом прочесть получившийся «суп» из многочисленных копий одной и той же ДНК.
Секвенирование по Сэнгеру
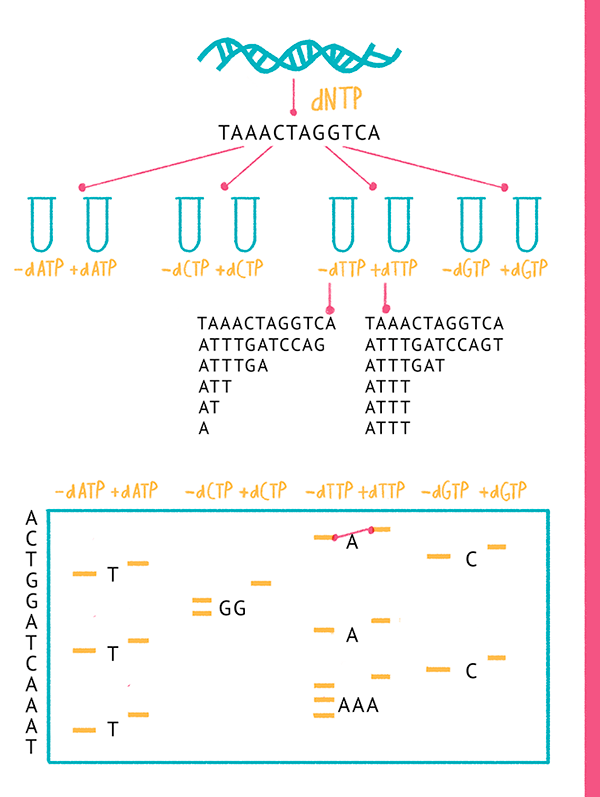
Первым методом секвенирования, который учёные сумели применить для обработки целых геномов (в том числе генома человека), стало секвенирование по Сэнгеру (Sanger sequencing). Смысл таков: участок ДНК клонируется, после чего полученная смесь делится на четыре части. Каждая часть помещается в активную среду, где присутствуют:
Собственно, процесс практически идентичен клонированию ДНК, с которым мы встретились в предыдущем разделе. Разница только в том, что теперь в один из нуклеотидов подмешаны «ложные» нуклеотиды; они могут образовать точно такую же водородную связь, но не могут продолжить свою нить дальше.
В результате в каждой части образуется большое число копий префиксов исследуемого участка ДНК, которые имеют разную длину, но всегда заканчиваются на одну и ту же букву – в зависимости от того, когда повезёт взять в процесс клонирования «ложный» нуклеотид. Например, в пробирке, где все последовательности заканчиваются на Т, из нашего примера выше получилась бы смесь из следующих префиксов:
ATGCAGAACAGACGATCAGCGACACTTTA (образец)
AT
ATGCAGAACAGACGAT
ATGCAGAACAGACGATCAGCGACACT
ATGCAGAACAGACGATCAGCGACACTT
ATGCAGAACAGACGATCAGCGACACTTT
Как теперь, получив такую смесь, «прочесть» геномную последовательность? Заметим, что в сумме в четырёх пробирках мы получили все возможные префиксы интересующего нас участка. Это значит, что если мы сможем просто измерить длину каждого префикса (точнее говоря, даже не измерить, а просто упорядочить, узнав, кто из них длиннее), то мы сможем узнать и последовательность тоже. Предположим, что мы увидели, что в пробирках лежат префиксы вот такой длины (по порядку, от самого лёгкого 1 до самого тяжёлого 10):A C G T
1, 5, 7, 8, 10 4, 9 3, 6 2
Очевидно, что эта последовательность начинается с А (т.к. самый лёгкий префикс, из одной буквы, заканчивается на A); дальше идёт C, дальше опять A, и так далее. В результате можно прочесть исходный участок: ATGCAGAACA.
А чтобы измерить длину, можно, например, измерить массу всех префиксов во всех пробирках. Чтобы измерить массу, можно, например (разные секвенаторы использовали разные процедуры, но суть от этого не меняется), ионизировать эти молекулы и отправить их наперегонки к заряженному электроду в специальном геле, который создаст трение и замедлит продвижение молекул – этот метод называется электрофорезом. При одинаковом заряде более тяжёлые молекулы будут двигаться медленнее, и в результате получится примерно такая картинка.

Рисунок из Википедии
Видно, что (в идеальном случае) можно просто прочесть последовательность нуклеотидов от самого лёгкого префикса (т.е. префикса из одной буквы) к самому тяжёлому.
Результаты и ошибки сэнгеровского секвенирования
На выходе из сэнгеровского секвенатора получаются короткие участки ДНК, так называемые риды (reads). Для биоинформатики принципиальны две вещи: во-первых, какой длины получаются риды, во-вторых, какие в них могут быть ошибки и как часто (разумеется, на свете нет ничего идеального).
Сэнгеровские риды по этим критериям очень хороши: получаются риды длиной около тысячи нуклеотидов, причём качество начинает заметно падать только после 700-800 нуклеотидов. Сам процесс секвенирования по Сэнгеру, с которым мы познакомились в предыдущем разделе, предопределяет и эффект падения качества (труднее отличить молекулу массой 700 от молекулы массой 701, чем массу 5 от массы 6), и другой неприятный эффект – если в геноме встречается длинная последовательность из одной и той же буквы (…AAAAAAAA…), трудно бывает точно определить, какой она длины – все промежуточные массы попадут в одну и ту же пробирку, некоторые из них могут не встретиться, некоторые слиться друг с другом и т.д. Но всё же сэнгеровское секвенирование даёт отличные результаты с достаточно длинными ридами, которые потом относительно легко собирать. О том, как это делается, мы будем говорить в последующих текстах.
Именно при помощи сэнгеровского секвенирования был впервые расшифрован геном человека. Секвенирование по Сэнгеру применяется и сегодня, но его всё активнее вытесняют другие методы, и применяется оно всё реже. Кому же и почему оно уступило свои позиции?
Секвенаторы второго поколения: Illumina
Современные секвенаторы – это так называемые секвенаторы второго поколения (SGS, second generation sequencing). В них участки ДНК по-прежнему многократно клонируются, но процесс чтения устроен не так, как у Сэнгера. Существует много разных методов, отличающихся довольно существенно, поэтому мы рассмотрим только один из них, один из самых популярных на сегодня – секвенирование по методу Solexa (ныне Illumina; в смене названия не нужно искать глубокий смысл, просто одна компания купила другую).
Процесс секвенирования Illumina проиллюстрирован на рисунке; кроме того, можно посмотреть один из нескольких существующих видеороликов с анимацией этого процесса – в данном случае, действительно, лучше один раз увидеть, чем сто раз прочесть текст. Однако краткие комментарии тоже пригодятся; вот как происходит процесс секвенирования по методу Illumina.
В результате на каждом цикле мы прочитываем одновременно очень большое число нуклеотидов из разных последовательностей. Но за это приходится платить тем, что участки ДНК, которые мы можем прочесть, оказываются гораздо короче, чем в случае секвенирования по Сэнгеру – риды Illumina обычно получаются длиной около 100 нуклеотидов.
Парные риды и постановка задачи
Есть ещё одна важная деталь. Участки ДНК «присасываются» к подложке обоими концами, причём мы можем узнать, какие последовательности соответствуют одному и тому же участку. Это значит, что в реальности мы читаем один и тот же участок, длина которого нам приблизительно известна, сразу с двух сторон. В результате данные получаются примерно такого вида:
причём расстояние между известными строчками (число вопросительных знаков) известно не совсем точно. В зависимости от технологии, можно получить как очень длинные неизвестные фрагменты (около 1000 нуклеотидов), «обрамлённые» двумя ридами длины 100, так и короткие фрагменты, в которых неизвестны буквально два-три десятка нуклеотидов между ридами. И те, и другие могут очень помочь в сборке, и об этом мы тоже будем говорить в следующих сериях.
Итак, теперь мы можем формально поставить задачу сборки геномов. Она звучит так: по большому числу подстрок небольшой длины восстановить исходную длинную строку в алфавите из букв A, C, G, T. В случае секвенирования по методу Illumina – по большому числу пар коротких подстрок, разделённых в исходной строке приблизительно известным расстоянием. Поставив эту задачу, мы можем забыть про биологию, химию и медицину – перед нами чисто алгоритмическая задача. Однако, прежде чем перейти к математике, сделаем ещё несколько замечаний.
Ошибки и показатели качества в секвенаторах второго поколения
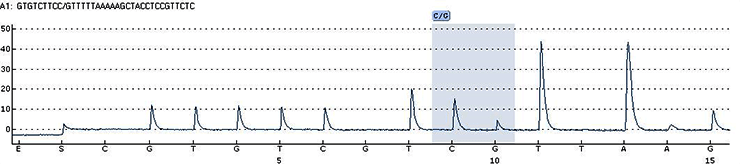
Как мы уже знаем, секвенирование всегда содержит ошибки. В секвенаторах Illumina и аналогичных ошибки, как правило, происходят на фазе, когда нужно распознать помеченные нуклеотиды, т.е. понять, каким цветом и с какой силой светятся кластеры из многократно клонированных участков ДНК. На рисунке – типичный пример такой фотографии, порождённой секвенатором Illumina.

Рисунок с сайта medicine.yale.edu
Проблема здесь заключается в том, что из-за неидеальности остальных этапов процесса кластеры никогда не светятся только одним цветом; это всегда смесь всех четырёх цветов с той или иной интенсивностью. Нужно выделить наиболее интенсивную компоненту и оценить, насколько вероятна ошибка в этой букве; эта задача называется base calling (распознавание нуклеотидов). Base calling – это целая наука, в подробности которой мы сейчас вдаваться не будем.
Для нас сейчас важно, что в результате каждому нуклеотиду каждого рида секвенатор ставит в соответствие вероятность того, что этот нуклеотид был распознан правильно. Эти вероятности тоже можно использовать при сборке, и секвенаторы выдают их вместе с собственно ридами.
В итоге типичный рид в так называемом fastq-формате, стандартном для секвенаторов второго поколения, выглядит примерно так:
@EAS20_8_6_1_3_25/1
GCAAAAAACTTACCCCGGAACAGGCCGAGCAGATCAAAACGCTACTGCAATACAGACCATCAAGCACCAACTCCCNNNCGTAGNNNNNNTATGTTNNNNG
+EAS20_8_6_1_3_25/1
HHHHHHHGHHHHHHHHHHHHHHHHHHHHEHHHHHHHHEGHHHHGHHGHEFD?A=A&FFBB>&::===@&@E@E>A#########################
Первая и третья строки содержат имя рида; вторая строка – сама последовательность нуклеотидов. Обратим внимание, что среди букв A, C, G, T встречаются и буквы N – это значит, что секвенатор не смог однозначно определить, какой здесь был нуклеотид, и сдался. А четвёртая строка кодирует, в логарифмическом масштабе, вероятности того, что тот или иной нуклеотид распознан правильно; например, H здесь соответствует вероятности ошибки около одной десятитысячной. Как правило, качество ухудшается к концу рида; в нашем примере, как видите, хвост рида и вовсе не удалось сколь-нибудь надёжно прочитать.
Другие методы секвенирования
Хотя мы подробнее всего рассмотрели секвенатор Illumina (Solexa), на самом деле на этом методе свет клином не сошёлся. Есть и другие секвенаторы второго поколения, с другими свойствами.
В секвенировании лигированием (sequencing by ligation) на фазе, когда уже нужно распознавать нуклеотиды, используют не ДНК-полимеразу и процесс репликации, а специальные короткие «зонды», которые присоединяются к комплементарным нуклеотидам, фиксируются, затем вымываются, и процесс повторяется снова. Так устроен секвенатор SOLiD от Applied Biosystems.
Пиросеквенирование (pyrosequencing) основано на хемилюминесцентных сигналах, которые подают специально модифицированные нуклеотиды, когда соединяются с комплементарным нуклеотидом в прочитываемой нити ДНК; на этом принципе работает, например, секвенатор 454 от 454 Life Sciences.
Принцип работы секвенатора PacBio (от Pacific Biosciences) очень похож на принцип работы Illumina, но у него по-другому устроен метод детектирования – специальные «решётки» позволяют уловить сигналы от отдельных молекул (метод получил название SMRT, single molecule real time sequencing). Это позволяет ускорить процесс, уместить больше ридов на одной подложке (нужно меньше клонировать ДНК, не нужно выращивать большие кластеры) и существенно увеличить длину надёжно прочитываемых ридов.
Недавно появившийся метод ионного полупроводникового секвенирования (на нём основан секвенатор IonTorrent) вместо всего этого просто детектирует соединения (ионы), которые выделяются при присоединении нового нуклеотида к нити ДНК. Это позволяет радикально сократить время и стоимость получаемых ридов, хотя процент ошибок становится больше, и больше становится ошибок в фрагментах из повторяющейся одной буквы.
Человеческая мысль не стоит на месте: методы секвенирования постоянно улучшаются. Однако практически все современные методы выдают относительно короткие риды, от 100 до 400 нуклеотидов; в этом цикле мы будем в основном говорить о том, как собирать именно короткие риды.
Sanger или Illumina?
Человеческий геном был впервые собран на сэнгеровских секвенаторах, причём алгоритмическая сторона того проекта была проработана гораздо меньше, чем сейчас, десять лет спустя. Алгоритмы, которыми собирали первый человеческий геном, значительно проще тех, о которых мы будем говорить в дальнейшем. Однако первый геном всё-таки собрали; может быть, весь алгоритмический прогресс – это никому не нужный миф, и вполне хватило бы старых программ?
Невероятно, но факт: «старые» секвенаторы (первого поколения, сэнгеровские) выдают значительно более подходящие для сборки данные, чем «новые» (второго поколения). Это в основном выражается в длине ридов (reads), тех участков ДНК, которые удаётся последовательно прочесть, и которые, собственно, и нужно собрать в одну большую строчку. Секвенаторы первого поколения выдавали риды длиной более пятисот нуклеотидов, обычно около тысячи. Современные секвенаторы выдают пары ридов, каждый из которых имеет длину около ста нуклеотидов.
На таком уровне становится важной и цена алгоритмической стороны вопроса. Чтобы сборка геномов не занимала дольше и не стоила дороже, чем само их секвенирование, нужно разработать очень быстрые алгоритмы для решения задачи сборки. Об этом пойдет речь в следующей статье.
12 методов в картинках: секвенирование нуклеиновых кислот
12 методов в картинках: секвенирование нуклеиновых кислот
Авторы
Редакторы
Секвенирование ДНК и РНК — это рутинный процесс, позволяющий, тем не менее, вникнуть в суть всего живого. Первоначально расшифровка генома была «развлечением» для избранных, а сегодня заказать эту услугу может каждая вторая научно-исследовательская лаборатория. С каждым годом проникнуть в дебри геномной, транскриптомной и эпигеномной информации становится все проще. Этот обзор посвящен основным принципам секвенирования нуклеиновых кислот и может послужить превосходным путеводителем как для любителя, изучающего основы молекулярной биологии, так и для специалиста, который планирует эксперимент и грезит научными прорывами.
12 биологических методов в картинках
Генеральный партнер цикла — компания «Диаэм»: крупнейший поставщик оборудования, реагентов и расходных материалов для биологических исследований и производств.
Партнер этой статьи — «СкайДжин»
Пять причин выбрать SkyGen:
Одна из главных миссий «Биомолекулы» — докопаться до самых корней. Мы не просто рассказываем, какие новые факты обнаружили исследователи — мы говорим о том, как они их обнаружили, стараемся объяснить принципы биологических методик. Как вытащить ген из одного организма и вставить в другой? Как проследить в огромной клетке за судьбой нескольких крошечных молекул? Как возбудить одну крохотную группу нейронов в огромном мозге?
И вот мы решили рассказать о лабораторных методах более системно, собрать воедино в одной рубрике самые главные, самые современные биологические методики. Чтоб было интереснее и нагляднее, мы густо проиллюстрировали статьи и даже кое-где добавили анимации. Мы хотим, чтобы статьи новой рубрики были интересны и понятны даже случайному прохожему. И с другой стороны — чтобы они были так подробны, что даже профессионал мог бы обнаружить в них что-то новое. Мы собрали методики в 12 больших групп и собираемся сделать на их основе биометодический календарь. Ждите обновлений!
Появление и развитие технологий секвенирования
С момента открытия нуклеиновых кислот прошло уже почти полторы сотни лет. В далеком 1869 году Иоганн Фридрих Мишер выделил из находившихся в гное клеток неизвестное доселе вещество, содержащее азот и фосфор, которое назвал нуклеином, а затем (из-за его свойств) — нуклеиновой кислотой. Первоначально считалось, что молекулы нуклеиновых кислот — резерв фосфора в клетках, однако уже в первой половине XX века ученые доказали их наследственную природу. Тогда же появилось понятие гена —наименьшей структурной и функциональной единицы наследственности, — и сформировалась новая наука — генетика.
Вплоть до середины прошлого века структура носителей генетической информации и способы ее передачи оставались неясными. Модель двойной спирали ДНК, которая входит во все современные учебники генетики и молекулярной биологии, предложили в 1953 году Френсис Крик и Джеймс Уотсон (за это в 1962 ученые получили Нобелевскую премию). Последовавшие следом открытие генетического кода и разработка центральной догмы молекулярной биологии дали мощный толчок к развитию естественных наук, в первую очередь — генетики. Основные исторические вехи этого процесса показаны на рисунке 1.
Рисунок 1. История геномных исследований и секвенирования нуклеиновых кислот. 8 марта 1865 г. — Грегор Мендель доложил результаты своих опытов, объясняющие механизм наследования. 1869 г. — Иоганн Фридрих Мишер выделил из клеток в гное неизвестное доселе вещество, содержащее азот и фосфор, которое назвал нуклеином. 1905–1909 гг. — появление терминов «ген» и «генетика». 1953 г. — Френсис Крик и Джеймс Уотсон предложили структуру двойной спирали ДНК. 1958 г. — Френсис Крик сформулировал центральную догму молекулярной биологии. 1975 г. — Сэнгер с коллегами разработал «плюс-минус» метод секвенирования ДНК. 1977 г. — группа Сэнгера разработала метод «терминаторов». 1977 г. — Максам и Гилберт предложили метод секвенирования ДНК путем химической деградации. 2001 г. — опубликован первый полный геном человека. 2005 г. — коммерциализация технологии пиросеквенирования. 2006 г. — коммерциализация технологии Solexa (Illumina). 2006 г. — коммерциализация технологии лигазного секвенирования. 2010 г. — коммерциализация технологии ионного полупроводникового секвенирования (технология PostLight TM ). 2011 г. — первый коммерческий релиз секвенаторов PacBio, основанных на технологии одномолекулярного SMRT-секвенирования. 2015 г. — начало продаж первых приборов, основанных на секвенировании через нанопору. Чтобы увидеть рисунок в полном размере, нажмите на него.
Осознав основные принципы функционирования нуклеиновых кислот (НК), научное сообщество предприняло грандиозные усилия для того чтобы разработать быстрые и эффективные методы определения их первичной последовательности (рис. 2) — как это принято говорить сегодня, секвенирования. Спустя десятилетия после открытия Уотсона и Крика в биологической науке наступила новая эпоха — эра секвенирования нуклеиновых кислот и геномики.
Рисунок 2. Секвенирование нуклеиновых кислот (другими словами, определение их нуклеотидной последовательности) — это расшифровка первичной структуры линейных молекул ДНК или РНК, состоящих из последовательности «букв» — нуклеозидтрифосфатов: дАТФ, дГТФ, дЦТФ, дТТФ и УТФ. Эти «буквы» зачастую именуют просто по азотистому основанию, входящему в их состав — аденин (А), гуанин (Г), цитозин (Ц), тимин (Т), а также урацил (У) в случае РНК.
Секвенирование белковых молекул
Кстати, секвенировать можно не только нуклеиновые кислоты, но и другие биополимеры — например, пептиды и белки, «сотканные» из аминокислотных остатков. Однако в этом спецпроекте мы предпочли подробно остановиться на секвенировании только нуклеиновых кислот и не затрагиваем отдельно тему белкового секвенирования.
Примечание от «Диаэм»: Белковое секвенирование методом Эдмана проводят на приборах Shimadzu в сочетании с ВЭЖХ.
Словарик
Почти четверть века назад в Соединенных Штатах стартовал грандиозный по своему масштабу научный проект, посвященный определению последовательности генома человека [1]. Основной его целью стала расшифровка генетической информации, заключенной в хромосомах (компактизованная ДНК), которые мы наследуем от своих родителей. В течение тринадцати лет многочисленные исследовательские группы по всему миру работали над определением полной последовательности генома человека. Почти три миллиарда долларов, потраченные на этот проект, открыли перед исследователями замечательные перспективы. Используя полученные данные, появилась возможность искать и находить участки ДНК, связанные с генетически обусловленными заболеваниями. И если природа многих моногенных болезней (вызываемых отказом единственного гена нашего генома) стала понятна уже давно, то некоторые заболевания — сердечно-сосудистые, онкологические, болезни Альцгеймера [2] и Паркинсона — являются многофакторными: вызвать их может широкий спектр изменений генома, многие из которых до сих пор неизвестны. Информация о генетических вариантах, связанных с человеческими недугами, позволяет формировать научно-обоснованный подход при их диагностике и лечении.
Расшифровка и аннотация (маркировка генов и других объектов в последовательности ДНК) генома человека поставили вопрос об использовании генетической информации как для диагностики заболеваний и их долговременного прогнозирования у человека, так и для исследования популяционной структуры сообществ, этногенеза и эволюционных процессов. Применение современных технологий секвенирования и генотипирования предлагает перспективные способы решения задач современной медицинской геномики и эпигеномики.
Такие результаты могут быть использованы при создании систем для проведения дифференциальной диагностики и выявления генетической природы заболеваний, для проведения персональной терапии и подбора методик лечения на основе анализа индивидуальных генетических характеристик. Решение таких задач тесно связано с разработкой эффективных алгоритмов и математических моделей для биоинформатической обработки данных геномного секвенирования и их использованием на базе суперкомпьютерных кластеров.
Геномные исследования позволяют решать массу задач как прикладного, так и фундаментального плана. Благодаря им разрабатываются новые лекарства и продукты, они же позволяют проникнуть в глубокую историю человечества [3] или понять причину массового вымирания видов.
Сейчас разработано несколько способов секвенирования НК. Самый популярный и надежный из них — секвенирование по Сэнгеру — позволяет «считывать» последовательности до 1000 пар оснований (п.о.) и используется для небольших фрагментов генома/генов или для валидации результатов более современного секвенирования нового поколения (next-generation sequencing, NGS), где размер одного прочитанного фрагмента варьирует от 25 до 500 п.о. В отличие от секвенирования по Сэнгеру, методы NGS используют для глубокого (многократного) прочтения генетического материала, которое необходимо, например, для ресеквенирования и сборки новых геномов (de novo), транскриптомных и эпигеномных исследований [4]. Помимо этого, NGS-секвенирование значительно производительнее, позволяя одновременно считывать миллионы и даже миллиарды коротких фрагментов. Такой рост производительности привел к возможности определения последовательности сразу десятков геномов (в зависимости от их размера) за один запуск прибора (рис. 3).
Рисунок 3. Современные технологии делают процесс секвенирования ДНК рутинной процедурой.
Стремительно развивающиеся новые технологии секвенирования ДНК позволяют быстро и эффективно определять особенности организмов на уровне их геномов. Главным итогом развития геномных и постгеномных технологий стало существенное расширение возможностей изучения генетической природы целого спектра заболеваний человека. Масштабные ассоциативные исследования на больших клинических выборках позволяют получать данные о генетических характеристиках, присущих конкретным группам людей (семьям, популяциям), развивая методы персонализированной медицины. В связи с этим, изучение механизмов генетической предрасположенности к многофакторным заболеваниям и выявление специфических генетических маркеров сегодня имеет особенную актуальность. Подобные методы широко применяются за рубежом и в России, где технологии современного секвенирования также постепенно внедряют в медицинские исследования и медицинскую практику с целью персонификации стратегии лечения [1].
Технологической основой для подобных исследовательских и сугубо прикладных проектов служат геномные секвенаторы (приборы, на которых проводят секвенирование), поставляемые различными коммерческими компаниями, такими как Illumina, Thermo Fisher Scientific, Oxford Nanopore Technologies, Pacific Biosciences и другие.
В 2017 году на рынке представлены сразу несколько перспективных разработок в области секвенирования НК. Эти подходы применены в секвенаторах нового поколения:
и некоторых других.
Современные технологии делают процесс секвенирования ДНК рутинной процедурой, особенно в том случае, когда речь идет об организмах с уже известной последовательностью генома — их последующая биоинформатическая обработка не представляет значительного труда, поскольку исследователь уже имеет референсный (ранее отсеквенированный) геном, который позволяет избежать ошибок при анализе полученных данных. При анализе нового, неопубликованного ранее, генома (de novo секвенирование и сборка) перед исследователем стоит ряд более сложных задач, в ходе решения которых он пытается сложить единичные фрагменты в цельную последовательность, задействуя многочисленные математические алгоритмы и суперкомпьютерные мощности [5], [6].
Однако наибольший интерес представляет не сама последовательность генома, а понимание того, как он функционирует: какие гены обеспечивают жизнедеятельность клетки, как происходит регуляция (включение или выключение генов) или какие генные пути начинают работать в ответ на стрессовые факторы.
Все современные секвенирующие платформы отличаются от метода секвенирования по Сэнгеру тем, что не требуют этапа клонирования фрагментов ДНК. Это экономит рабочее время и позволяет избежать ряда проблем с клонированием АТ-богатых участков. Общий принцип пробоподготовки для большинства современных (NGS) секвенаторов включает фрагментирование ДНК, привязку к субстрату, амплификацию фрагментов с помощью ПЦР (в одномолекулярном секвенировании от ПЦР удалось отказаться) и последующее считывании последовательности НК. В отличие от метода секвенирования по Сэнгеру, современные платформы обеспечивают параллельное проведение миллиардов реакций в малых объемах, что позволяет получить намного больший объем информации на выходе.
Рассмотрим основные технологии секвенирования нуклеиновых кислот подробнее.
Секвенирование по Сэнгеру
«Плюс-минус» метод секвенирования ДНК
Один из наиболее популярных методов секвенирования обязан своим появлением английскому биофизику Фредерику Сэнгеру (1918–2013) — единственному ученому в истории мировой науки, получившему сразу две Нобелевские премии по химии (в 1958 и 1980 годах). Первую премию присудили за установление структур белков, особенно инсулина, а вторую награду ему вручили в том числе и за разработку методов определения первичной последовательности нуклеиновых кислот.
Методику секвенирования ДНК с использованием радиоактивно меченых нуклеотидов и ДНК-полимеразы (или фрагмента Кленова ДНК-полимеразы I) предложили Сэнгер и его коллеги в 1977 году, причем с течением времени этот метод прошел несколько модификаций и к настоящему моменту считается золотым стандартом современного секвенирования.
Первоначально Ф. Сэнгер и Алан Коулсон разработали так называемый «плюс-минус» метод секвенирования ДНК [7], который можно подразделить на две основные стадии:
Рисунок 4. «Плюс-минус» метод секвенирования ДНК, предложенный Ф. Сэнгером и А. Коулсоном.
Секвенирование ДНК по Сэнгеру: метод «терминаторов»
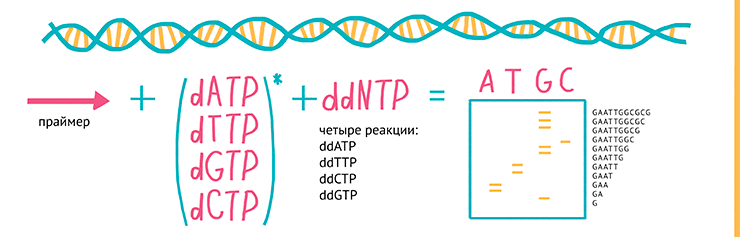
Спустя пару лет Сэнгер с коллегами предложил еще один способ секвенирования, получивший название метода «терминаторов» или метода «обрыва цепи» [9]. Суть этого метода заключается в том, что в реакционную смесь добавляют аналоги привычных нуклеотидов (дидезоксинуклеозидтрифосфаты), включение которых в синтезируемую цепь приводит к невозможности ее дальнейшего синтеза (терминации), а по образовавшемуся «обломку» можно установить последнюю букву секвенируемого фрагмента ДНК (рис. 5).
Рисунок 5. Метод «терминаторов»: используют ДНК-полимеразу, олигонуклеотидные праймеры и смесь четырех дезоксинуклеотидов (dNTPs) (А, Т, G и C), один из которых радиоактивно помечен по α-положению фосфата ( 32 P). В каждую из четырех реакций добавляется по одному 2’,3’-дидезоксинуклеозидтрифосфату (ddATP, ddTTP, ddCTP или ddGTP), которые терминируют дальнейшую реакцию (синтез комплементарной молекулы ДНК с матрицы) — таким образом в каждой пробирке образуется набор фрагментов ДНК разной длины, которые заканчиваются одним и тем же нуклеотидом. Затем полученные фрагменты визуализируют с помощью электрофореза и, сравнивая длины фрагментов из четырех реакций с ddATP, ddTTP, ddCTP или ddGTP, восстанавливают последовательность ДНК.
Автоматизированные модификации метода «терминаторов» активно применяют до сих пор в специальных приборах — секвенаторах. Открытие многочисленных флуоресцентных молекул позволило отказаться от использования радиоактивной метки и сделало возможным проведение реакции в одной пробирке. Реакционную смесь разделяют капиллярным электрофорезом, a выстроившиеся в синтезируемую цепочку ДНК меченые нуклеотиды затем регистрируют детекторами флуоресценции, предоставляя возможность считывать последовательность всего секвенируемого ДНК-фрагмента.
Секвенаторы по Сэнгеру: решения от «Диаэм»
Капиллярные секвенаторы ДНК Applied Biosystems (Thermo Fisher Scientific) — золотой стандарт секвенирования по Сэнгеру. Производительность — 4, 8, 24, 48 и 96 образцов. Они предназначены для секвенирования относительно небольших фрагментов ДНК длиной до 1000 нуклеотидов, характеризуются высокой точностью и используются для генотипирования человека, животных и других организмов (в том числе в медицине — трансплантологии, перинатологии, онкологии, фармакогенетике и др.), для выявления точечных мутаций, делеций и инсерций, для валидации мутаций, выявленных NGS-секвенированием, для анализа метилирования ДНК, для идентификации личности в криминалистике и др.
Новейшая разработка в этой области — капиллярный секвенатор SeqStudio (Applied Biosystems):
Первые впечатления пользователей нового капиллярного секвенатора SeqStudio
Материал предоставлен партнёром — компанией «Диаэм»
Другие методы секвенирования «старого поколения»
Кроме методов, предложенных Сэнгером, в конце прошлого века развивались и другие подходы к определению последовательности НК, которые — в частности, метод химической деградации, разработанный Максамом и Гилбертом [10], — не получили дальнейшего распространения из-за быстрого развития энзимологии (раздел биохимии, изучающий ферменты), которая предоставила преимущество методу «терминаторов» Сэнгера.
Использование секвенирования по Сэнгеру
Секвенирование по Сэнгеру позволяет «считывать» последовательности до 1000 пар нуклеотидов и применяется для небольших фрагментов генома/генов. В частности, оно используется для:
Наиболее популярными секвенаторами, использующими технологию секвенирования по Сэнгеру, являются приборы, производимые компанией Thermo Fisher Scientific: 3730xL, 3730, 3500xL, 3500, 3130xL, 3130, 310.
Следует отметить, что все описанные выше типы исследований сейчас можно проводить с помощью секвенирования «нового поколения», речь о котором пойдет в следующей главе, однако главные плюсы секвенирования по Сэнгеру — высокая точность (достоверность) полученных данных и невысокая стоимость работ при анализе небольшого количества ДНК-фрагментов — сохраняют актуальность этого типа определения последовательности НК.
Секвенирование «нового поколения» — next-generation sequencing (NGS)
За последние полтора десятилетия были разработаны, коммерциализированы и продолжают успешно развиваться совершенно новые технологии определения последовательности НК, в основе которых лежит стремление к миниатюризации, автоматизации, увеличению объема получаемых данных, а также удешевлению процесса. Появление NGS впервые позволило значительно ускорить и удешевить определение полной последовательности миллионов геномов организмов, начиная от бактерий и заканчивая человеком. Более того, появилась реальная возможность единовременно оценивать экспрессию (работу) тысяч генов в организмах, тканях и единичных клетках (секвенирование транскриптомов), а также анализировать регуляцию их активности (анализ экспрессии микроРНК и метилирования генома). В настоящее время на рынке представлено сразу несколько разработок, позволяющих определять последовательность полных геномов организмов, проводить анализ экспрессии генов и метилирования генома. Эти подходы реализуются на секвенаторах нового поколения производства коммерческих компаний Illumina, Thermo Fisher Scientific, Pacific Biosciences и Oxford Nanopore Technologies. Часть разработанных платформ уже ушли с рынка (например, GS FLX, 454/Roche или HeliScope/Helicos Bioscience); другие, пройдя несколько реинкарнаций и модификаций, прочно закрепились на нем (Illumina и Thermo Fisher Scientific); третьи только нащупывают почву, намереваясь занять свою нишу и найти своего потребителя (например, Oxford Nanopore Technologies).
Появление высокопроизводительных технологий секвенирования сопровождается прогрессом программного обеспечения — создаются алгоритмы с открытым программным кодом, появляются открытые источники данных и платформы для вычислений. Новые математические и информационные технологии позволяют геномике развиваться быстрее и использовать более сложные алгоритмы. Эти алгоритмы могут включать в себя сразу несколько приложений и программ и позволяют работать с очень большим объемом данных.
Секвенирование «нового поколения» применяется как для анализа геномов организмов, для которых уже доступен референсный геном (ресеквенирование), так и для того, чтобы впервые расшифровать геном организма (секвенирование de novo).
Для ресеквенирования успешно используют платформы, генерирующие большое количество коротких чтений (секвенируемых фрагментов ДНК), поскольку даже относительно короткие фрагменты ДНК успешно картируются (картирование, или выравнивание, — процесс биоинформатического поиска расположения конкретного короткого фрагмента в полной геномной последовательности) на референсный геном (последовательность ДНК в цифровом виде, составленную учеными как общий репрезентативный пример последовательности генома конкретного вида) при биоинформатическом анализе данных. Такие выравненные чтения могут использоваться для поиска однонуклеотидных полиморфизмов (SNPs), малых делеций и инсерций или других структурных изменений в геноме.
Что касается секвенирования de novo и сборки новых, ранее не прочитанных, геномов, то использование коротких чтений сильно усложняет сборку, особенно в случае больших по размеру и сложно устроенных геномов эукариот (например, полиплоидных геномов). В этих случаях используют комбинированный подход — сочетание платформ, генерирующих как короткие, так и длинные чтения. При сборке генома de novo ученые уподобляются ребенку, пытаясь правильно сложить элементы геномного пазла (короткие фрагменты отсеквенированной ДНК) в единую картину, созданную эволюцией за сотни миллионов лет (рис. 6).
Рисунок 6. Cеквенирование de novo и сборка новых, ранее непрочитанных, геномов требует значительных усилий.
В то же время наибольший интерес представляет отнюдь не сама последовательность генома, а понимание того, как он функционирует. Новые методы секвенирования НК позволяют оценивать уровень метилирования ДНК, проводить анализ дифференциальной экспрессии генов, в том числе и генов-регуляторов (например, микроРНК). Возможность анализа экспрессии генов в биологических системах открывает перед исследователем огромные возможности. Например, этот метод можно применять при исследовании функционирования центральной нервной системы человека (для понимания основных молекулярных аспектов работы головного мозга [11], [12]), при оценке защитного ответа клеток на атаки вирусов [13] или ответной реакции на стресс [14]. Не менее интересные данные может дать изучение регуляции экспрессии генов посредством анализа их метилирования [15] или изучение экспрессии некодирующих РНК [16], [17].
Оценка уровня метилирования генома, например, позволяет определить, какие генные пути и сети включаются в ответ на меняющиеся факторы окружающей среды; такие работы зачастую проводят для изучения эволюционных механизмов в живых системах. Изучение экспрессии кодирующих белки и некодирующих РНК в разных тканях и клетках также позволяет понять и описать гены, вовлеченные в жизнедеятельность клеток, органов и организмов.
Пиросеквенирование
Технологию предложил в 1996 году Пол Нирен с коллегами из Королевского технологического института в Стокгольме [19]. Затем ее коммерциализировали (2005 год) и воплотили в приборе GS FLX, 454 производства Roche (2008 год). Этим методом можно определять нуклеотидную последовательность фрагментов геномной ДНК размером 300–500 пар оснований (п.о.). Особо следует отметить тот факт, что подавляющее большинство NGS-методов требуют предварительной фрагментации ДНК для упрощения ферментативных реакций. К обоим концам фрагментированной ДНК «пришивают» ДНК-адаптеры (данная конструкция называется ДНК-библиотекой), необходимые для эмульсионной ПЦР (эПЦР) на магнитных сферах и последующего секвенирования.
Готовые ДНК-библиотеки иммобилизуют на магнитных сферах. Затем магнитные сферы с нанесенной на них клональной библиотекой доставляют на проточную ячейку, где в присутствии праймера, дезоксинуклеотидтрифосфатов и ферментов — ДНК-полимеразы, люциферазы, АТФ-сульфурилазы — происходит циклический синтез новой цепи.
Во время цикла пиросеквенирования при образовании фосфодиэфирной связи между матричной цепочкой ДНК и нуклеотидом синтезируемой цепи выделяется пирофосфат, который запускает каскад химических реакций, приводящих к выделению АТФ, необходимой для реакции окисления люциферина с выделением кванта света, который фиксируют аналоговой интегральной микросхемой (ПЗС-матрицей), состоящей из светочувствительных фотодиодов. Нуклеотиды, не вовлеченные в синтез новой цепи, удаляют из проточной ячейки, и начинается следующий реакционный цикл, в ходе которого добавляют дезоксинуклеотидтрифосфат другого типа (рис. 7).
Рисунок 7. Принцип пиросеквенирования. При включении нуклеотида в синтезируемую цепочку ДНК происходит регистрация высвобождающихся пирофосфатов — побочного продукта реакции полимеризации нуклеотидов в ДНК.
Пиросеквенирование от «Диаэм»
Принцип пиросеквенирования используется в секвенаторе PyroMark Q24, производимом компанией Qiagen. Этот прибор предназначен для одновременного прочтения 24 коротких последовательностей (до 100 нуклеотидов) и широко применяется в научных исследованиях и диагностике для точечного генотипирования и анализа метилирования. Основное достоинство секвенатора PyroMark — высокая точность и возможность анализа практически любого точечного полиморфизма.
Материал предоставлен партнёром — компанией «Диаэм»
Технология секвенирования на молекулярных кластерах с использованием флуоресцентно меченых нуклеотидов
Технология секвенирования на молекулярных кластерах, так же как и пиросеквенирование, подразумевает синтез новой молекулы ДНК по матрице. Этот метод начали разрабатывать еще в середине 90-х годов прошлого века химики Шанкар Баласубраманиан и Дэвид Кленерман из Кембриджа, изучавшие работу ДНК-полимеразы на молекулярном уровне, используя флуоресцентно меченые нуклеотиды и ДНК-матрицу, иммобилизованную на поверхности [20]. Творческие семинары в лаборатории и дружеские посиделки в баре привели к коммерциализации этой технологии в 2006 году под брендом Solexa, который спустя год был приобретен компанией Illumina. Сейчас платформа продолжает развиваться, и потребителям предлагают новые линейки приборов.
Суть метода заключается в следующем. К обоим концам предварительно фрагментированной ДНК лигируют адаптеры, необходимые для ПЦР и последующего секвенирования на молекулярных кластерах. Полученные ДНК-библиотеки иммобилизуют на поверхности проточной ячейки, где и проводят циклический процесс секвенирования. Реакционная смесь для синтеза комплементарной ДНК подается на поверхность проточной ячейки и содержит ферменты, олигонуклеотиды, а также четыре типа флуоресцентно меченых дезоксинуклеозидтрифосфатов. После включения в синтезируемую цепь ДНК нуклеотида-терминатора идентифицируют с помощью ПЗС-матрицы как тип включенного нуклеотида, так и его положение. Затем терминирующая группа и флуоресцентная краска отщепляются от нуклеотида, и цикл синтеза повторяется. Эта серия шагов продолжается определенное количество раз, число которых задает пользователь (рис. 8).
Рисунок 8. Принцип секвенирования на молекулярных кластерах: полимеризация (синтез «дочерней» цепочки) ДНК с использованием флуоресцентно меченых нуклеотидов внутри специальной камеры, регистрирующей флуоресценцию.
Размер чтений, получаемых с секвенатора, может достигать 300 п.о. (прибор Illumina MiSeq). Кроме того, серия секвенаторов 2017 года NovaSeq позволяет определять последовательность до 48 геномов человека за один запуск прибора.
Технология циклического лигазного секвенирования
Технология циклического лигазного секвенирования была разработана группой Джорджа Макдональда Черча [21] и, в отличие от представленных выше, использует метод лигирования (формирование химических связей между нуклеотидами при помощи специального фермента — лигазы). Данный подход к секвенированию НК коммерциализировали в 2006 году, и приборы, известные под брендом SOLiD, уже длительное время представлены на рынке (первоначально развитием этой системы секвенирования занималась компания Applied Biosystems, а затем Life Technologies и Thermo Fisher Scientific).
Суть метода заключается в определении нуклеотидной последовательности фрагментов геномной ДНК размером 25–75 п.о. К обоим концам предварительно фрагментированной ДНК лигируют адаптеры, необходимые для эПЦР на магнитных сферах и последующего секвенирования на проточной ячейке.
Магнитные сферы с нанесенной на них клональной библиотекой помещают на проточную ячейку, где и происходит секвенирование с помощью лигирования восьминуклеотидных зондов, несущих четыре различных флуорофора на 5’-конце. Флуоресценция считывается с помощью специальной камеры после каждого цикла секвенирования и, затем переводится в последовательность нуклеотидов (рис. 9).
Рисунок 9. Принцип лигазного секвенирования и последовательность зондов, которые используют при секвенировании. Зонды для секвенирования можно разделить на несколько участков (начиная с 3’-конца): первые два нуклеотида зонда лигируются к комплементарным участкам на секвенируемой ДНК-библиотеке, нуклеотиды с третьего по пятый — вырожденные (т.е. могут гибридизоваться с любыми тремя нуклеотидами секвенируемой ДНК-библиотеки). Нуклеотиды с шестого по восьмой также способны гибридизоваться с любыми тремя нуклеотидами секвенируемой ДНК-библиотеки, однако они отщепляются вместе с флуоресцентным красителем в конце каждого цикла секвенирования.
Технология лигазного секвенирования применялась в приборах, выпускаемых под брендом SOLiD. Спустя несколько поколений приборов пробоподготовку усовершенствовали, и на рынок вышла новая линейка приборов — 5500, 5500xl, а также 5500w, использующий изотермальную ПЦР (WildFire технология) для клонирования ДНК-библиотек [22].
Ионное полупроводниковое секвенирование
Технология, предлагаемая в этой приборной линейке, основана на использовании полупроводниковых микрочипов для секвенирования. Суть этого подхода весьма проста и заключается в регистрации локального изменения рН на микрочипе в момент удлинения синтезируемой цепи ДНК-полимеразой на ДНК-матрице (рис. 10).
Рисунок 10. Принцип полупроводникового секвенирования основан на обнаружении ионов водорода, которые выделяются во время полимеризации ДНК.
Секвенаторы нового поколения от «Диаэм»
Высокопроизводительные секвенаторы нового поколения Ion S5/S5 XL (Thermo Fisher Scientific) работают по технологии полупроводникового секвенирования Ion Torrent, которая обеспечивает простейшую процедуру таргетного секвенирования, высокую скорость и экономичность. Использование микрочипов различной производительности позволяет проводить на одном приборе анализ как отдельных генов, бактериальных геномов, так и экзомов и транскриптомов.
Материал предоставлен партнёром — компанией «Диаэм»
Пробоподготовка (приготовление ДНК-библиотек) напоминает таковую при циклическом лигазном секвенировании. Первоначально ДНК фрагментируют, затем к концам полученных фрагментов лигируют специфические ДНК-адаптеры, необходимые для эмульсионной ПЦР на магнитных сферах и последующего секвенирования.
Подготовка библиотек для NGS: решение от «Диаэм»
Выделение НК и подготовка библиотек для NGS — это довольно трудоемкий процесс, который включает множество шагов, занимает немало времени и требует максимальной сосредоточенности оператора. На помощь ученым-лаборантам приходят автоматизированные технологии на каждом из этапов пробоподготовки:
Материал предоставлен партнёром — компанией «Диаэм»
Одномолекулярное секвенирование
SMRT-секвенирование (single molecule real time sequencing), предложенное сотрудниками компании Pacific Biosciences, не только позволило отказаться от проведения полимеразной цепной реакции при пробоподготовке, но и дало возможность наблюдать за работой ДНК-полимеразы, наращивающей синтезируемую цепь, в реальном времени [23].
Создание платформы Pacific Biosciences не только решило проблему ПЦР-дупликатов, но и значительно увеличило длину чтений, что крайне важно при сборке геномов de novo. Суть метода заключается в определении нуклеотидной последовательности фрагментов геномной ДНК размером до 20 000 п.о. с лигированными к их концам специфическими ДНК-адаптерами, необходимыми для последующего секвенирования.
Сама реакция секвенирования молекул ДНК проходит в специальных ячейках (SMRT-ячейки) на прозрачной (кремниевой) подложке, с напыленным на нее слоем алюминия. В основе метода лежит использование технологии Zero-mode waveguide (ZMW) [24]. Сквозь дно в ячейку подается свет, однако благодаря особенностям ее строения, пучок фотонов не рассеивается, а освещает только конкретную часть (на подложке), где закреплена молекула phi29 ДНК-полимеразы. Эта полимераза была выбрана в качестве «считывающего» фермента благодаря своей высокой точности, скорости синтезирования дочерней цепи и эффективной работе с нуклеотидами, несущими флуоресцентную метку [25].
Смысл SMRT-секвенирования схож с описанными ранее методами NGS — ДНК-полимераза достраивает вторую цепь исследуемой молекулы ДНК, используя нуклеотиды, меченные различными флуоресцентными метками, которые регистрируют при помощи конфокальной микроскопии. В 2016 году анонсировали покупку платформы Pacific Biosciences биотехнологическим гигантом Roche Diagnostics, однако сделка так и не состоялась [26].
Разработка другого способа одномолекулярного секвенирования (коммерциализированного к настоящему времени) началась в конце прошлого века, когда группа американских ученых наглядно продемонстрировала возможность побуждать молекулы ДНК и РНК проходить сквозь ионный канал диаметром 2,6 нм в двуслойной липидной мембране под воздействием электрического поля. Более того, уже тогда исследователи сумели различать ДНК и РНК, а также оценивать длину входящих в нанопору олигонуклеотидов [27]. Спустя 13 лет впервые продемонстрировали возможность определения последовательности НК нанопоровым секвенированием [28], а затем данную технологию коммерциализировала и представила на рынок компания Oxford Nanopore Technologies (рис. 11).
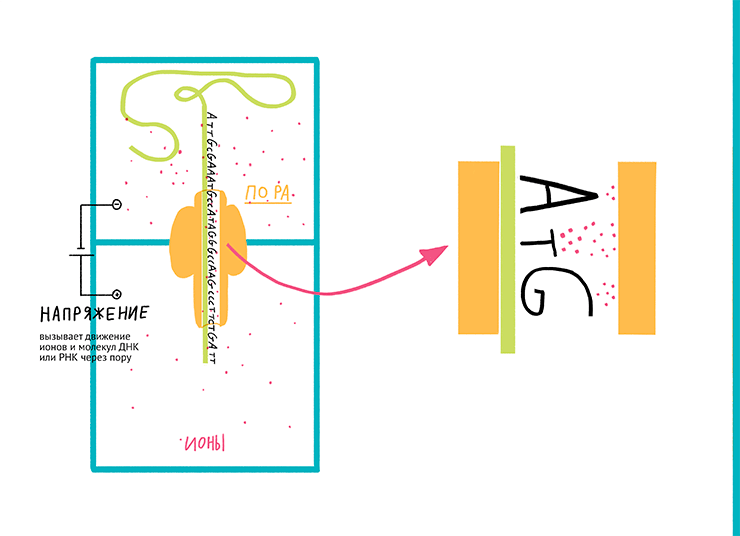
Рисунок 11. Принцип нанопорового секвенирования, коммерциализованный компанией Oxford Nanopore Technologies. Данный тип секвенирования основан на измерении меняющейся силы тока при прохождении молекулы НК сквозь нанопору в двухслойной мембране.
Суть работы нанопоровых систем (MinION, GridION X5, PromethION и SmidgION), предложенных британской компанией, достаточно проста. Реакционная камера, в которой проходит процесс считывания последовательности НК, разделена двухслойной мембраной с единичной порой. К камере прикладывается напряжение, вызывающее движение ионов и молекул ДНК или РНК через пору. При прохождении молекулы НК сечение поры (доступное для миграции ионов) уменьшается, в результате чего сила тока падает. Таким образом, считывая изменение силы тока, можно определять тип нуклеотида, проходящего через пору в конкретный отрезок времени.
Кроме описанных выше двух технологий компания Helicos Biosсiences пыталась продвинуть на биотехнологический рынок свою технологию одномолекулярного секвенирования — true Single Molecule Sequencing (tSMS). Данный подход во многом схож с технологией секвенирования на молекулярных кластерах (Illumina), однако позволяет обходиться без ПЦР при пробоподготовке.
Суть метода заключается в определении нуклеотидной последовательности фрагментов геномной ДНК размером до 50 п.о. К обоим концам предварительно фрагментированной ДНК лигируют адаптеры. Полученные ДНК-библиотеки иммобилизуют на поверхности проточной ячейки, где и проводят циклический процесс секвенирования. Один цикл состоит из удлинения синтезируемой на матрице цепочки за счет одного из четырех флуоресцентно меченых нуклеотидов, присоединение которого детектируется прибором.
Небольшая длина чтения (до 50 п.о., медиана — 30 п.о.) и большое количество ошибок (3–5%) данной платформы привели к оттоку потенциальных покупателей. Технология не получила широкого распространения, несмотря на потенциальную применимость в работе со сложными образцами, например, в палеогеномике [29], где отказ от ПЦР мог бы привести к сокращению процентного соотношение экзогенной (мусорной) ДНК в анализируемых образцах древней ДНК [30]. В итоге, в конце 2012 года компания Helicos Biosciences была признана банкротом и прекратила свое существование [31].
Одномолекулярное секвенирование длинных фрагментов ДНК может найти свое применение в самых разнообразных областях, и, что самое интересное, при работе с единичными клетками позволяет описывать их молекулярный «портрет» [32]. Это особенно важно при анализе транскриптомов клеток, позволяя описывать все возможные изоформы активных генов — благодаря длине чтений, недоступных для технологии Solexa или PostLight [33].
Секвенирование третьего поколения
Технологию одномолекулярного секвенирования можно также назвать технологией третьего поколения, поскольку она работает по принципу, совершенно отличному от того, который применяется в секвенаторах второго поколения. Кроме того, характерной особенностью таких секвенаторов является их сверхкомпактность. Например, самый компактный портативный секвенатор MiniION имеет вес всего 100 граммов и работает от USB 3.0 обычного компьютера или ноутбука. Первые приборы MiniION уже применяют и в России (официальный дистрибьютор Oxford Nanopore — компания «Диаэм»).
MiniION — портативный прибор для прямого секвенирования ДНК/РНК онлайн от Oxford Nanopore.
Для высокопроизводительного секвенирования Oxford Nanopore разработал приборы, в которые можно параллельно установить несколько секвенаторов MiniION:
В настоящее время Oxford Nanopore работает над созданием модели секвенатора SmidgION, который будет работать от обычного смартфона и сможет применяться в полевых условиях, где нет лаборатории и даже компьютера.
Материал предоставлен партнёром — компанией «Диаэм»
Партнер этой публикации — компания SkyGen: молодой игрок на рынке продукции для LifeScience-лабораторий в России и СНГ
У нас есть три основных принципа:
Бóльшая часть производителей в нашем портфолио — это прямые эксклюзивные поставщики. Мы являемся первым звеном в поставках для таких компаний как Agilent Technologies, New England Biolabs, Oxford Nanopore, QIAGEN, Macherey-Nagel, NIMAGEN, EdgeBio, Thermo Scientific, SIGMA-ALDRICH, Santa Cruz Biotechnology, ChemGenes, Bruker, Dornier, Bio Molecular Systems, Gilson, Opentrons, Titertek-Berthold.
В 2017 году компания «СкайДжин» стала официальным дистрибьютором нанопоровых секвенаторов Oxford Nanopore Technologies в России, Белоруссии и Казахстане. Наши специалисты окажут техническую поддержку и проведут обучение на месте. С нами технология нанопорового секвенирования стала доступной для вас!
Мы верим: чтобы быть близкими к вам, недостаточно просто выставлять счета и осуществлять доставку. Нам важно глубоко разобраться в теме и быть поддержкой для вас. Для этого мы публикуем новости, обзоры и высоко ценим любые замечания и критику. Каждый продукт мы «пропускаем» через себя. Анализируем плюсы и минусы, представляем, как и где он будет работать, консультируемся с вами. И, как результат, создаем методички, проводим семинары и презентации.
Сегодня мы предлагаем современные решения для проведения секвенирования на различных платформах — от капиллярного секвенирования и NGS до мономолекулярного анализа с технологией Oxford Nanopore. Выбор за вами!
Ознакомиться с потенциалом применения новой технологии нанопорового секвенирования MinION можно здесь.
Вы всегда можете обратиться к нам, связавшись с менеджерами или написав по адресу info@skygen.com, а также позвонив по телефону +7 (495) 215-02-22 или бесплатной линии 8 (800) 333-12-26.
Мы стараемся угнаться за вами в научном познании мира, мы стараемся учиться и расти вместе с вами!
Материал предоставлен партнёром — компанией «СкайДжин»
Использование секвенирования нового поколения
Производительность и относительная доступность NGS-методов привели к настоящей революции в биологической и медицинской науке. Более того, благодаря новым подходам появилась реальная возможность проводить ранее технически недоступные исследования. Использование секвенирования нового поколения позволяет проводить такие проекты как:
HLA-типирование с помощью NGS-методик
Примером практического применения таргетного секвенирования в медицинских целях может быть HLA-типирование — секвенирование генов главного комплекса гистосовместимости, ответственных за распознавание организмом клеток по типу «свой-чужой». HLA-типирование проводят при трансплантации, при патологиях беременности, при аутоиммунных заболеваниях и др. NGS, в отличие от других методов HLA-типирования (серологический анализ, методы SSP и SSO, секвенирование по Сэнгеру), позволяет получить полную последовательность генов главного комплекса и гистосовместимости и, соответственно, точный генотип.
Набор для HLA-типирования Holotype HLA от компании Omixon позволяет не только секвенировать эти гены, но и правильно интерпретировать результаты секвенирования. Этот продукт включает в себя реагенты для пробоподготовки и программное обеспечение HLA Twin для интерпретации и предназначен для всех современных моделей NGS-секвенаторов.
Материал предоставлен партнёром — компанией «Диаэм»
Решение по анализу транскриптома от «Диаэм»
Большим недостатком метода NGS является высокая стоимость реагентов. Однако на сегодняшний день можно найти на рынке относительно недорогую альтернативу: например, наборы Lexogen для анализа транскриптома. Это хорошее решение для полнотранскриптомного секвенирования, построения экспрессионного профиля, обогащения фракции мРНК и удаления нецелевых фракций РНК. Для интерпретации данных секвенирования хорошим помощником является программное обеспечение RNA-Seq от Lexogen.
Материал предоставлен партнёром — компанией «Диаэм»
Заключение
В наши дни происходит бурное развитие технологий, связанных с исследованием генов, белков и других молекулярных структур живых организмов. Разрабатываются портативные, быстрые, точные и универсальные методы исследований биологических объектов. Появление высокопроизводительных технологий секвенирования НК сопровождается развитием в области программного обеспечения, создаются алгоритмы с открытым программным кодом. Новые математические и информационные технологии позволяют геномике развиваться быстрее и использовать более сложные алгоритмы (например, нейронные сети).
На сегодняшний день объемы получаемой секвенаторами информации значительно обогнали возможности математического анализа получаемых результатов. Но даже несмотря на это, биомедицинская наука вовлечена в круговорот геномной революции, когда новые данные появляются ежедневно, а биотехнологические компании предлагают все новые и новые решения, значительно облегчающие диагностику заболеваний, приближая мир к новому направлению — персонализированной медицине [34].
Календарь
На основе статей спецпроекта мы решили сделать календарь «12 методов биологии» на 2019 год. Эта статья представляет апрель.
 Геномика: постановка задачи и методы секвенирования
Геномика: постановка задачи и методы секвенирования